El ciclo celular es un conjunto ordenado de sucesos que conducen al crecimiento de la célula y la división en dos células hijas. Las células que no están en división no se considera que estén en el ciclo celular. Las etapas, mostradas a la derecha, son G1-S-G2 y M. El estado G1 quiere decir "GAP 1"(Intervalo 1). El estado S representa "Síntesis". Este es el estado cuando ocurre la replicación del ADN. El estado G2 representa "GAP 2"(Intervalo 2). El estado M representa «la fase M», y agrupa a la mitosis (reparto de material genético nuclear) y citocinesis (división del citoplasma). Las células que se encuentran en el ciclo celular se denominan «proliferantes» y las que se encuentran en fase G0 se llaman células quiescentes.1 Todas las células se originan únicamente de otra existente con anterioridad.2 El ciclo celular se inicia en el instante en que aparece una nueva célula, descendiente de otra que se divide, y termina en el momento en que dicha célula, por división subsiguiente, origina dos nuevas células hijas.


FASES DEL CICLO CELULAR:
La
célula puede encontrarse en dos estados claramente diferenciados:
3- El estado de no división o interfase. La célula realiza sus funciones específicas y, si está destinada a avanzar a la división celular, comienza por realizar la duplicación de su ADN.
- El estado de división, llamado fase M.
- Interfase
Es el período comprendido entre divisiones celulares. Es la fase más larga del ciclo celular, ocupando casi el 90% del ciclo, trascurre entre dos mitosis y comprende tres etapas:
4- Fase G1 (del inglés Growth o Gap 1): Es la primera fase del ciclo celular, en la que existe crecimiento celular con síntesis de proteínas y de ARN. Es el período que trascurre entre el fin de una mitosis y el inicio de la síntesis de ADN. Tiene una duración de entre 6 y 12 horas, y durante este tiempo la célula duplica su tamaño y masa debido a la continua síntesis de todos sus componentes, como resultado de la expresión de los genes que codifican las proteínas responsables de su fenotipoparticular. En cuanto a carga genética, en humanos (diploides) son 2n 2c.
- Fase S (del inglés Synthesis): Es la segunda fase del ciclo, en la que se produce la replicación o síntesis del ADN, como resultado cada cromosoma se duplica y queda formado por doscromátidas idénticas. Con la duplicación del ADN, el núcleo contiene el doble de proteínas nucleares y de ADN que al principio. Tiene una duración de unos 6-8 horas.
- Fase G2 (del inglés Growth o Gap 2): Es la tercera fase de crecimiento del ciclo celular en la que continúa la síntesis de proteínas y ARN. Al final de este período se observa al microscopio cambios en la estructura celular, que indican el principio de la división celular. Tiene una duración entre 3 y 4 horas. Termina cuando la cromatina empieza a condensarse al inicio de la mitosis. La carga genética de humanos es 2n 4c, ya que se han duplicado el material genético, teniendo ahora dos cromátidas cada uno.
- Fase M (mitosis y citocinesis)
Es la división celular en la que una célula progenitora (células eucariotas, células somáticas -células comunes del cuerpo-) se divide en dos células hijas idénticas. Esta fase incluye la
mitosis, a su vez dividida en:
profase,
metafase,
anafase,
telofase; y la
citocinesis, que se inicia ya en la telofase mitótica. Si el ciclo completo durara 24 h, la fase M duraría alrededor de media hora (30 minutos).
REGULACIÓN DEL CICLO CELULAR:
La regulación del ciclo celular, explicada en el año 2001 en organismos eucariotas,
5 puede contemplarse desde la perspectiva de la toma de decisiones en puntos críticos, especialmente en la mitosis.
6 De este modo, se plantean algunas preguntas:
1- ¿Cómo se replica el ADN una única vez? Una pregunta interesante es cómo se mantiene la euploidía celular. Sucede que, en la fase G1, la Cdk(ciclina) promueve la adición al complejo de reconocimiento del origen de replicación del ADN de unos reguladores llamados Cdc6, los cuales reclutan a Mcm, formando un complejo prerreplicativo del ADN, que recluta a la maquinaria dereplicación genética. Una vez que se inicia la fase S, la Cdk-S produce la disociación de Cdc6 y su posterior proteólisis, así como la exportación al citosol de Mcm, con lo que el origen de replicación no puede, hasta el ciclo siguiente, reclutar un complejo prerreplicativo (las degradaciones proteolíticas siempren conllevan irreversibilidad, hasta que el ciclo gire). Durante G2 y M se mantiene la unicidad de la estructura de prerreplicación, hasta que, tras la mitosis, el nivel de actividad Cdk caiga y se permita la adición de Cdc6 y Mdm para el ciclo siguiente.
- ¿Cómo se entra en mitosis? La ciclina B, típica en la Cdk-M, existe en todo el ciclo celular. Sucede que la Cdk (ciclina) está habitualmente inhibida por fosforilación mediante la proteína Wee, pero, a finales de G2, se activa una fosfatasa llamada Cdc25 que elimina el fosfato inhibidor y permite el aumento de su actividad. Cdk-M inhibe a Wee y activa a Cdc25, lo que produce unaretroalimentación positiva que permite la acumulación de Cdk-M.
- ¿Cómo se separan las cromátidas hermanas? Ya en mitosis, tras la formación del huso acromático y superación del punto de restricción de unión a cinetocoros, las cromátidas han de eliminar su esqueleto de cohesinas, que las unen. Para ello, Cdk-M favorece la activación de APC, una ligasa de ubiquitina, por unión a Cdc20. Esta APC ubiquitiniza y favorece la ulterior degradación en el proteasoma de la segurina, inhibidor del enzima separasa que debe escindir las cohesinas.

Metafase tardía: placa metafásica previa a la separación de las cromátidas.
- ¿Cómo se sale de mitosis? Una vez que los niveles de Cdk-M son altos, parece difícil detener la dinámica de mitosis y entrar en citocinesis: pues bien, esto ocurre porque la APC activada por la Cdk-M, y tras un lapso cuyo mecanismo de control es aún desconocido, ubiquitiniza a la ciclina B, produciendo el cese absoluto de actividad Cdk-M.
- ¿Como se mantiene el estado G1? En la fase G1, la actividad Cdk está muy disminuida porque: APC-Hct1 (Cdc20 sólo actúa en mitosis) elimina toda ciclina B; se acumulan inhibidores de Cdk; la transcripción de ciclinas se ve disminuida. Para escapar de este reposo, se deben acumular ciclinas de G1. Esto se controla mediante factores de proliferación celular, señales externas. Los mecanismos moleculares de activación de transcripción de genes de las fases S y G2 necesarios para proseguir el ciclo son apasionantes: éstos genes están regulados por la proteína reguladora E2F, la cual se une apromotores de ciclinas G1/S y S. E2F está controlada por la proteína del retinoblastoma (Rb), la cual, en ausencia de factores tróficos, inhibe la actividad promotora de la transcripción de E2F. Cuando existen señales de proliferación, Cdk-G1 fosforila Rb, que pierde afinidad por E2F, se disocia de éste y permite que se expresen los genes de la fase S. Además, como E2F acelera la transcripción de su propio gen, las Cdk-S y G1/S fosforilan también a Rb y a Hct1 (activador de APC, que degradaría estas ciclinas), se produce una retroalimentación positiva.
[editar]Componentes reguladores
El ciclo celular es controlado por un sistema que vigila cada paso realizado. En regiones concretas del ciclo, la célula comprueba que se cumplan las condiciones para pasar a la etapa siguiente: de este modo, si no se cumplen estas condiciones, el ciclo se detiene.
1 Existen cuatro transiciones principales:
Los genes que regulan el ciclo celular se dividen en tres grandes grupos:
7- Genes que codifican proteínas para el ciclo: enzimas y precursores de la síntesis de ADN, enzimas para la síntesis y ensamblaje de tubulina, etc.
- Genes que codifican proteínas que regulan positivamente el ciclo: también llamados protooncogenes.8 Las proteínas que codifican activan la proliferación celular, para que células quiescentes pasen a la fase S y entren en división. Algunos de estos genes codifican las proteínas del sistema de ciclinas y quinasas dependientes de ciclina. Pueden ser:
- Genes de respuesta temprana, inducidos a los 15 minutos del tratamiento con factores de crecimiento, sin necesidad de síntesis proteica;
- Genes de respuesta tardía, inducidos más de una hora después del tratamiento con factores de crecimiento, su inducción parece estar causada por las proteínas producidas por los genes de respuesta temprana.
- Genes que codifican proteínas que regulan negativamente el ciclo:También llamados genes supresores tumorales.
Los
protooncogenes son genes cuya presencia o activación a oncogenes pueden estimular el desarrollo de cancer. cuando se activan exageradamente en las células normales provocan que ellas pierdan el control de la división y se mantengan proliferando sin control.

Expresión diferencial de ciclinas en las distintas fases del ciclo.
Las
ciclinas son un grupo heterogéneo de proteínas con una masa de 36 a 87 kDa. Se distinguen según el momento del ciclo en el que actúan.
1 Las ciclinas son proteínas de vida muy corta: tras disociarse de sus kinasas asociadas, se degradan con extrema rapidez.
Las
kinasas dependientes de ciclinas (CDK por sus siglas en
inglés) son moléculas de mediano
peso molecular que presentan una
estructura proteica característica, consistente en dos lóbulos entre los cuales está el
centro catalítico, donde se inserta el
ATP (que será el donador de
grupos fosfato.
9 En el canal de entrada al centro catalítico existe una
treonina que debe estar fosforilada para que la quinasa actúe. No obstante, en el propio centro hay dos treoninas que, al ser fosforiladas, inhiben a la quinasa y una región de unión a la ciclina llamada
PSTAIRE.
4 Existe una tercera región en las CDK, alejada del centro catalítico, a la que se une la proteína CKS, que regula la actividad kinasa de la CDK.
Relación del algunas ciclinas de vertebrados y levaduras1
| Vertebrados | | Levaduras | |
| Complejo Cdk/ciclina | Ciclina | Cdk asociada | Ciclina | Cdk asociada |
| Cdk-G1 | ciclina D | Cdk 4,6 | Cln3 | Cdk1 |
| Cdk-G1/S | ciclina E | Cdk2 | Cln1,2 | Cdk1 |
| Cdk-S | ciclina A | Cdk2 | Clb5,6 | Cdk1 |
| Cdk-M | ciclina B | Cdk1 | Clb1,2,3,4 | Cdk1 |
[editar]Regulación de los complejos ciclina/CDK
Existen multitud de proteínas que modulan la actividad del complejo ciclina/CDK.
4 Como vías de activación, se conoce que el complejo ciclina A/CDK2 activa la proteína CAK, quinasa activadora de CDK, y la proteína CAK fosforila a la CDK, activándola. En cambio, la fosfatasa PP2a desfosforila a la CDK, inactivándola. A su vez, hay descritos complejos inhibidores CKI como la
p27 y
p21 que se unen a la ciclina y a la CDK al mismo tiempo bloqueando el sitio activo.
Las
enzimas ligasas de ubiquitina conducen a la ubiquitinación de las ciclinas, lo que las marca para su degradación en el
proteasoma y, por tanto, destruye la funcionalidad del complejo con la CDK. Una enzima ligasa de ubiquitina implicada en este proceso de regulación del ciclo celular es el
complejo SCF, que actúa sobre las ciclinas G
1/S. Otro complejo denominado APC (del inglés
anaphase promoting complex) actúa sobre ciclinas M.
1- Ciclinas G1 y G1/S: Durante G1,la proteína Rb (retinoblastoma) está unida a la proteína E2F, que a su vez está unida al ADN promotor de genes necesarios para la entrada en S. Al acumularse ciclinas de G1, los complejos ciclina G1/CDK fosforilan a Rb, que se inactiva y deja de inactivar a E2F. La actividad de E2F permite la transcripción de genes para la fase S. Se forman entonces complejos ciclina G1S/CDK y ciclina S/CDK, que inactivan más unidades de Rb, favoreciendo todavía más la actividad de E2F.
- Ciclinas S: El complejo ciclina S/CDK promueve la actividad de la ADN polimerasa y de otras proteínas de la replicación. EL complejo multiproteico ORC (del inglés origin recognition complex) está asociado al origen de replicación del ADN. En G1 forma el complejo prerreplicativo al asociarse a la proteína CDC6 y al anillo proteico MCM. Las MCM actúan como helicasas promoviendo la replicación. El complejo ciclina S/CDK también fosforila la CDC6, dejándola accesible para la ubiquitinación por SCF. Así evita una nueva replicación.
- Ciclinas M: El complejo ciclina M/CDK activado por CAK está presente en todo el ciclo, pero está inhibido por la quinasa WEE1, que la fosforila. Al final de G2 la fosfatasa CDC25 desfosforila la CDK y activa el complejo ciclina M/CDK.El complejo ciclina M/CDK fosforila varias proteínas durante la mitosis:
- El complejo CDC20/APC ubiquitina las ciclinas M para salir de la fase M.
- Genes supresores de tumores: Los genes supresores de tumores regulan negativamente el ciclo. Se encargan de que la mitosis no continúe si se ha producido una alteración del proceso normal. Entre estos genes, también llamados 'de verificación', se encuentran los que codifican:
- productos que evitan mutaciones de genes reguladores del ciclo
- proteínas que inactivan las CDK por fosforilación/desfosforilación (ej. quinasa WEE1, fosfatasa CDC25)
- proteínas CKI inhibidoras del ciclo (por ejemplo, p53,10 p21, p16)
- proteína Rb (proteína del retinoblastoma), cuya alteración génica recesiva causa el cáncer de retina con ese nombre.
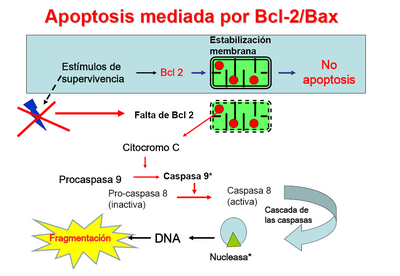
- proteínas que inducen la salida del ciclo hacia un estado celular diferenciado o hacia apoptosis (ej. Bad, Bax, Bak, receptor de ligando de Fas)
- La verificación se lleva a cabo en los puntos de control y asegura la fidelidad de la replicación y segregación del genoma. Algunos componentes, además de detectar fallos, pueden poner en marcha la reparación.
[editar]Puntos de control
Existen unos
puntos de control en el ciclo que aseguran la progresión sin fallos de éste, evaluando el correcto avance de procesos críticos en el ciclo, como son la replicación del ADN o la
segregación de cromosomas.
11 Estas rutas de verificación presentan dos características, y es que son
transitorias (desaparecen una vez resuelto el problema que las puso en marcha) y que pueden
caducar si el problema no es resuelto al cabo de un tiempo. Dichos puntos de control son:
1- Punto de control de ADN no replicado, ubicado al final de G1 antes de iniciar la fase S. Actúa inhibiendo a Cdc25, el cual es un activador de la Ciclina A/B Cdk1.
- Punto de control de ensamblaje del huso (checkpoint de mitosis), antes de la anafase. Se activa una proteína Mad2 que impide la degradación de la segurina, lo que impide la segregación de lascromátidas hermanas hasta que todas se hayan unido al huso. Es pues el punto de control de la separación de cromosomas, al final de la mitosis. En caso de que fuera incorrecto, se impediría la degradación de la ciclina B por parte de APC.
- Punto de control del daño del ADN, en G1, S o G2. El daño celular activa a p53, proteína que favorece la reparación del ADN, detiene el ciclo promoviendo la transcripción de p21, inhibidor de Cdk, y, en el caso de que todo falle, estimula la apoptosis.10
CICLO CELULAR Y CÁNCER:
Se cree que muchos tumores son el resultado de una multitud de pasos, de los que una alteración mutagénica no reparada del ADN podría ser el primer paso. Las alteraciones resultantes hacen que las células inicien un proceso de proliferación descontrolada e invadan tejidos normales. El desarrollo de un tumor maligno requiere de muchas transformaciones genéticas. La alteración genética progresa, reduciendo cada vez más la capacidad de respuesta de las células al mecanismo normal regulador del ciclo.8 Los genes que participan de la carcinogénesis resultan de la transformación de los genes normalmente implicados en el control del ciclo celular, la reparación de daños en el ADN y la adherencia entre células vecinas. Para que la célula se transforme en neoplásica se requieren, al menos, 2 mutaciones: una en un gen supresor de tumores y otra en un protooncogén, que dé lugar, entonces, a un oncogén
CICLO CELULAR EN PLANTAS:
Los programas de desarrollo en
plantas, a diferencia de lo que ocurre en animales, suceden tras la
embriogénesis. La proliferación y división celular está circunscrita a los
meristemos, zonas en las cuales se producen abundantes divisiones celulares que dan lugar a la aparición de nuevos órganos. Las
hojas y las
flores derivan del
meristemo apical del tallo y del
meristemo floral, respectivamente, mientras que el
meristemo radicular da lugar a la
raíz. La regulación, por tanto, de los programas de desarrollo se basa en buena medida en la
expresión génica particular de los meristemos y de la pauta concomitante de división celular; en plantas no existe la migración celular como mecanismo de desarrollo. La interacción antagonística entre las hormonas
auxina y
citoquinina parece ser el mecanismo clave para el establecimiento de identidades y pautas de proliferación durante la embriogénesis
12 y durante el desarrollo de los meristemos caulinar y radicular.
13El ciclo celular de plantas comparte elementos comunes con el de animales, así como ciertas particularidades. Las kinasas dependientes de ciclina (CDK) regulan, en buena medida, las características del ciclo celular. De este modo, CDKA (un equivalente a PSTAIRE CDK de animales), interviene en las transiciones G1/S y G2/M. No obstante, existen unas CDKB, únicas de plantas, que se acumulan en las fases G2 y M e intervienen en la transición G2/M.
En cuanto a ciclinas, las plantas poseen una diversidad mayor que los animales:
Arabidopsis thaliana contiene como mínimo 32 cilinas, quizá debido a los eventos de duplicación de su genoma.
14 La expresión de las diferentes ciclinas parece estar regulada por diversas
fitohormonas.
15- Ciclinas D: regulan la transición G1/S
- Ciclinas A: intervienen en el control de la fases S y M
- Ciclinas B: iimplicadas en las transiciones G2/M y en el control dentro de la fase M
- Ciclina H: parte de la kinasa activadora de CDKs.
Existe un complejo proteín
ligasa de ubiquitina semejante a APC/C (el
complejo promotor de la anafase)
16 y algunas ciclinas, como las de tipo B, poseen en su estructura secuencias de destrucción mediadas por ubiquitina: es decir, el proceso de
proteólisis es también una pieza clave en la regulación del ciclo celular en el mundo vegetal.
La fosforilación de complejos ciclina/CDK en el extremo
N terminal del elemento CDK inhibe la actividad del complejo; a diferencia de lo que sucede en animales, donde esta modificación postranscripcional sucede en residuos
Tyr o
Thr, en plantas sólo se da en los Tyr. En animales, la enzima que cataliza esta reacción es una WEE1 kinasa, y la fosfatasa, CDC25; en plantas existe un homólogo para WEE1, pero no para CDC25, que sí se ha encontrado en algas unicelulares.
17En cuanto a las proteínas inhibidoras de los complejos CDK/ciclina, se han descrito elementos similares a la familia Kip/Cip de mamíferos; concretamente, en plantas estos elementos inhibidores están modulados por la presencia de hormonas como la auxina o el ácido abscísico.
18 Estos y otros fitorreguladores desempeñan un papel clave en el mantenimiento de la capacidad meristemática y otros caracteres del desarrollo; ello depende de su concentración en una determinada zona y del programa de expresión génica presente en aquél lugar. Por ejemplo, las áreas que expresan a la proteína relacionada con el transporte de auxinas PINFORMED1 poseen una alta concentración de esta fitohormona lo que se traduce en la localización especial del que será el promordio de la futura hoja; al mismo tiempo, esto excluye la expresión de
SHOOTMERISTEMLESS, gen implicado en el mantenimiento de un estado indiferenciado de células meristemáticas madre (de lenta división).
19La vía del
retinoblastoma (vía RB/E2F/DP) no sólo se encuentra en animales y plantas, sino que también aparece en flagelados como
Chlamydomonas.
20 Un homólogo del supresor de tumores humano, denominado RETINBLASTOMA RELATED1, descrito en
A. thaliana, regula la proliferación celular en los meriestemos; está regulado vía fosforlización por parte de kinasas dependientes de ciclina.
21Un característica de gran flexibilidad de las células vegetales es la permisibilidad frente a endorreduplicaciones, esto es, duplicaciones de la dotación cromosómica (cambios de
ploidía), que se deben a la replicación del contenido genético sin que medie una
citocinesis. Este mecanismo es usual en determinados tejidos y organismos pero también puede suceder en plantas completas. Debido a que suele ir asociado a un mayor tamaño celular, ha sido objeto de selección en la mejora vegetal. Este hecho se explica debido al carácter sésil de los organismos vegetales y, por tanto, la imposibilidad de ejecutar comportamientos de evitación frente a estreses ambientales; de este modo, las plantas estresadas con un mayor número de copias del genoma podrían ser más resistentes. Los datos experimentales no siempre apoyan esta hipótesis.
22